Usando datos a largo plazo sobre enterovirus, un grupo de investigadores británicos desarrolló un modelo que puede predecir con exactitud la dinámica de transmisión de varias cepas y tipos diferentes de virus.
Los resultados podrían emplearse para poner en marcha programas de vacunación en los momentos más adecuados, lo que maximizaría la eficacia de dichos programas.
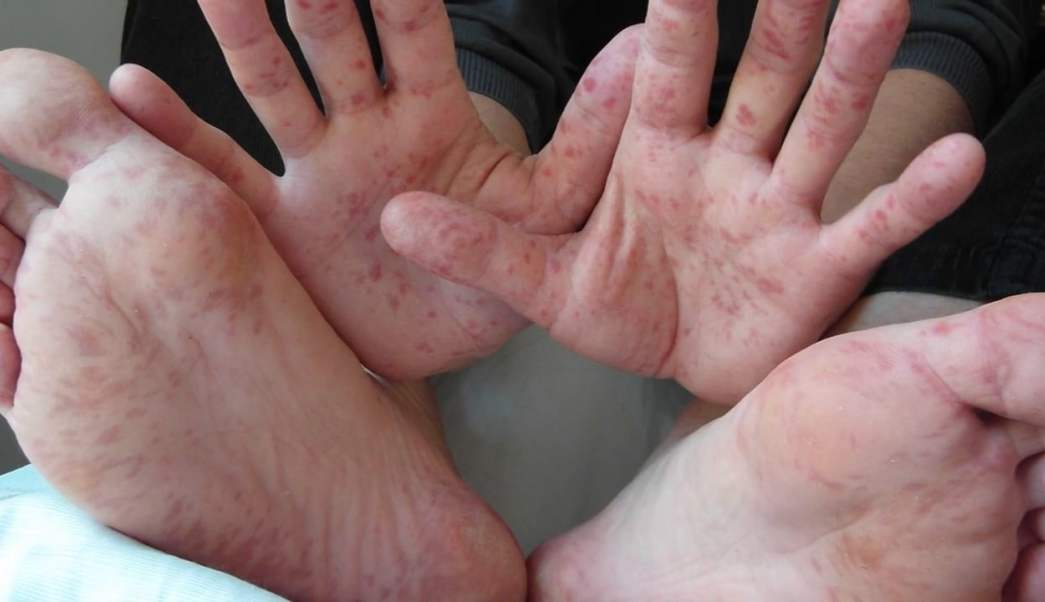
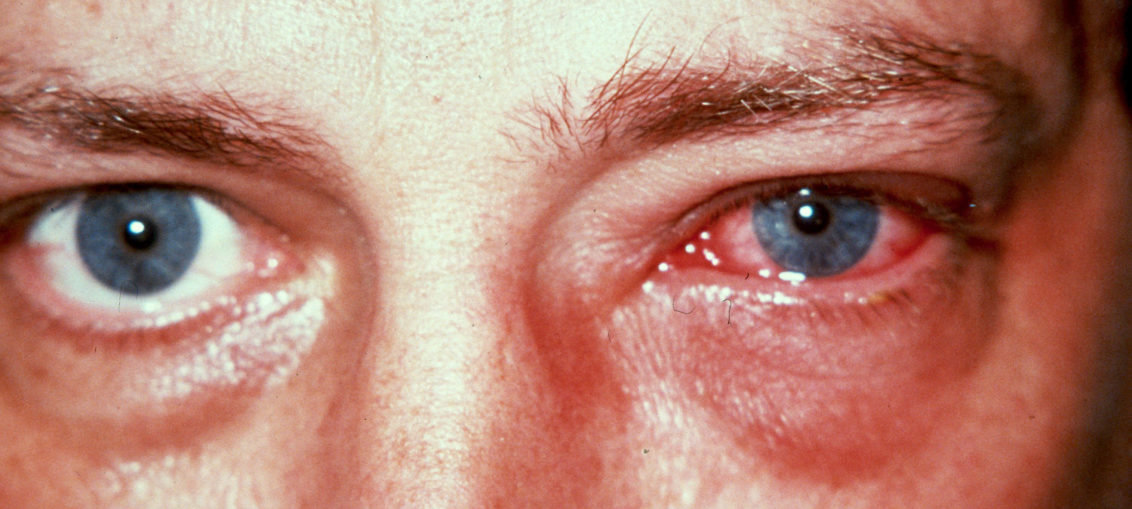
Más de 100 serotipos de enterovirus infectan a los humanos y provocan una amplia gama de enfermedades, entre las que se encuentran la meningitis, la encefalitis, la parálisis, las infecciones de las vías respiratorias superiores y la fiebre aftosa humana, entre otras.
En este estudio, Margarita Pons Salort y Nicholas Grassly, del Imperial College de Londres, analizaron datos obtenidos a través de la vigilancia a largo plazo de enterovirus recopilados de 2000 a 2015 en Japón, uno de los pocos países que cuenta con datos detallados sobre varias cepas y tipos de enterovirus, subraya un artículo sobre el tema, que gana espacio en la prensa especializada.
Los autores encontraron patrones donde, por ejemplo, una cepa de Coxsackievirus pasó de tener un ciclo de un año de 2000 a 2004 a tener un ciclo de dos años a partir de 2004, patrón que Pons-Salort y Grassly sospecharon que podría ser debido a la disminución general de nacimientos que ha habido en Japón en los últimos tiempos.
Usando estos datos, los investigadores desarrollaron un modelo único que refleja con gran precisión la dinámica de transmisión de 18 virus, de un total de 20.
En un estudio de Perspective relacionado, Birgit Nikolay y Simon Cauchemez escriben:
«Este análisis indica que podría ser posible predecir un brote de un serotipo de enterovirus específico, sin tener que dar cuenta de la protección cruzada conferida por otros serotipos en circulación. En el futuro, se podrían usar modelos para anticipar cuándo podría ocurrir el próximo brote de un enterovirus, sus proporciones y qué serotipo podría causarlo».
